(Für Literatur siehe Publikationen)
• Angewandtes
Antikörper-Engineering
Ausgehend von unserer Grundlagentechnologie zur Produktion
funktioneller Antikörperfragmente in E. coli (Skerra &
Plückthun 1988; Skerra, 1993) beschäftigen wir uns mit dem
Engineering von Antikörpern für interessante medizinische
(z.B. Tumortherapie, Virusinfektionen, neuronale Regeneration) und
biotechnologischen Anwendungen (Peptide Tags; neuartige Haptene;
Kokristallisation mit Proteinantigenen). Wir klonieren geeignete
Antikörper aus Hybridom-Zellinien oder führen Gensynthesen
durch, vor allem auch zum Zweck der "Humanisierung" von
Antikörpern (Schlapschy et al., 2004). Daneben beschäftigen
wir uns mit dem rationalen de novo Design von Antikörpern.
• Struktur-Funktionsanalyse von
Lipocalin-Proteinen
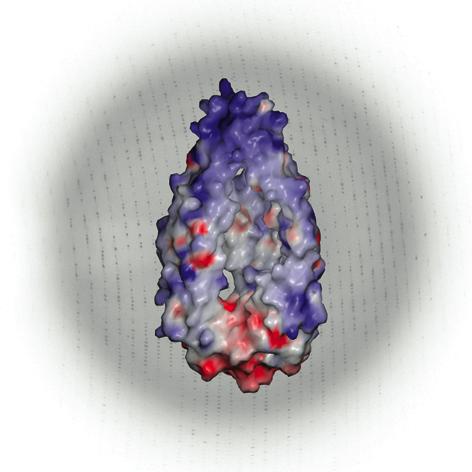
Die Lipocaline bilden eine weitverbreitete Klasse von Proteinen, von
Bakterien bis zum menschlichen Organismus, wo sie für den
Transport oder die Komplexierung von schlecht löslichen Vitaminen,
Hormonen oder chemisch empfindlichen metabolischen Verbindungen dienen.
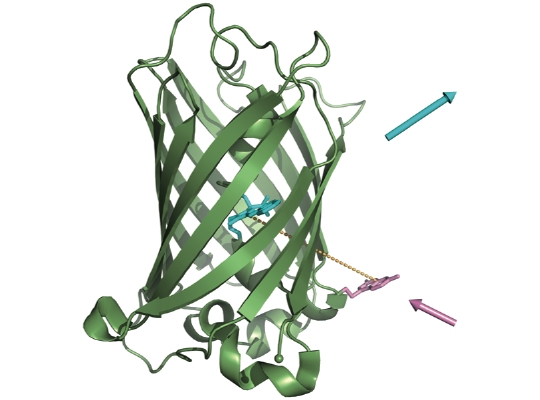
Sie weisen eine charakteristische Faltung mit einem zentralen
achtsträngigen β-Barrel und einer daran angelehnten α-Helix
auf. Die kelchförmige Faßstruktur ist an einem Ende zum
Lösungsmittel offen, wo vier Peptidschleifen den Eingang zur
Ligandenbindungstasche bilden. Wir beschäftigen uns mit dem
Studium der biochemischen Funktion vor allem der menschlichen Vertreter
dieser Proteinfamilie sowie mit deren systematischer Strukturanalyse.
So konnten wir unter anderem erstmals die Kristallstruktur des
Tränenlipocalins sowie des Apolipoprotein D (Eichinger et al.,
2007), eines Lipocalins im Blutplasma, aufklären.
•
Entwicklung der
ANTICALIN-Technologie
Während die faßartige Molekülarchitektur der Lipocaline
strukturell hochkonserviert ist, weist deren Bindungsstelle hohe
Variabilität auf, was im Einklang mit der Erkennung ihrer
unterschiedlichen natürlichen Liganden steht. Wir haben dieses
Prinzip aufgegriffen, um künstliche Lipocaline mit
maßgeschneiderten Erkennungseigenschaften herzustellen. Dazu
unterziehen wir die vier Peptidschleifen am Eingang zur Bindungstasche
einer lokalen Zufallsmutagenese, stellen so Proteinbibliotheken mit
großer kombinatorischer Komplexität her (> 1.000.000.000)
und isolieren daraus durch Phage Display und Colony Screening
Varianten, welche vorgegebene Liganden spezifisch und mit hoher
Affinität erkennen. Diese Klasse künstlicher Bindungsproteine
haben wir als Anticaline bezeichnet (Skerra & Schlehuber, 2005).
Nachdem wir ursprünglich mit kleinen Liganden begonnen haben
(Fluorescein, Digoxigenin), konzentrieren wir uns in der letzten Zeit
vor allem auf medizinisch relevante Proteintargets (CD4, Aβ usw.).
Die im Jahr 2001 gegründete Pieris
AG entwickelt und vermarktet die Anticalin-Technologie für
Anwendungen in der Humantherapie.
• Struktur-Funktionsanalyse von
Proteinen mit Relevanz in der Neuro-, Immun- und Entwicklungsbiologie
Wir beschäftigen uns mit Proteinen – häufig Membranrezeptoren
– wie Nogo-A (Zander et al., 2007), L1, MOG, Dkk-1, Calprotectin, CD33,
CD16, Langerin (Chatwell et al., 2007) usw. mit dem Ziel der
Röntgen-Strukturanalyse. Häufig gelingt es erst durch
Protein-Engineering, die für die Strukturanalyse relevanten
globulären Domänen einzugrenzen und deren gentechnische
Produktion in ausreichender Ausbeute und proteinchemischer Reinheit zu
realisieren. Dabei interessiert uns auch die Wechselwirkung dieser
Proteine mit Liganden oder mit Antikörperfragmenten. Viele dieser
Projekte werden in Kooperation mit anderen Arbeitsgruppen bearbeitet.
• Proteinstabilität und -faltung
sowie Chaperons
Für viele unserer Projekte ist die Analyse und auch das gezielte
Engineering der Stabilität von Proteinen von großer
Bedeutung. Diesbezüglich verfügen wir über die
notwendige apparative Ausstattung. Zudem interessiert uns die Effizienz
der intrazellulären Proteinfaltung, vor allem mit dem Ziel, diese
für wertvolle rekombinante Proteine zu optimieren. In diesem
Zusammenhang konnten wir die Kristallstruktur des periplasmatischen
Chaperons Skp von E. coli aufklären (Korndörfer et al.,
2004). Auch an der Struktur bestimmter eukaryontischer Chaperons (z.B.
BiP) sind wir interessiert. Zudem haben wir kürzlich ein
nützliches Koexpressionssystem für vier periplasmatische
Faltungshelferproteine in E. coli entwickelt (pTUM4; Schlapschy et al.,
2006).
• Manipulation pharmakologischer
Eigenschaften
Für die Wirkung vieler therapeutisch interessanter Proteine
(Antikörperfragmente, Interferone, Anticaline usw.) ist eine lange
Plasma-Halbwertszeit essentiell. Wir arbeiten diesbezüglich an
neuen Strategien. Z.B. haben wir eine bakterielle
Albuminbindungsdomäne zur Verlägerung der Zirkulation
eingesetzt, und wir sind dabei, neue Aminosäurepolymersequenzen zu
entwickeln, die sich mit dem therapeutischen Protein gentechnisch
fusionieren lassen und dessen Plasmastabilität deutlich
erhöhen (Schlapschy et al., 2007).
• Erweiterung des genetischen Codes
Der Einbau nichtkanonischer Aminosäuren mit neuartigen
Seitenketten und biochemischen Funktionen in rekombinante Proteine
besitzt großes Potential für die zukünftige Entwicklung
des Protein-Engineerings. Wir sind dabei, geeignete gentechnische
Systeme für die Biosynthese solcher modifizierter Proteine in E.
coli zu entwickeln, die nicht nur robust funktionieren sondern auch die
Produktion entsprechender Wirkstoffe im präparativen Maßstab
gestatten.
• Enzym-Engineering
Künstliche Enzyme mit neuen Substrat- und Produktspezifitäten
sowie verbesserter Stabilität sind nicht nur interessante
Studienobjekte für das rationale und kombinatorische
Protein-Design sondern spielen eine zunehmende Rolle für
Anwendungen in der "weißen" Biotechnologie. Wir haben
Forschungsprojekte begonnen, bei denen wir einerseits pflanzliche
Enzyme mit interessanten katalytischen Aktivitäten strukturell
charakterisieren und andererseits für die Industrie relevante
Enzyme mit evolutiven Methoden optimieren wollen.
• Methoden zur Produktion
rekombinanter Proteine
Wir haben in den vergangenen Jahren eine Reihe von Systemen entwickelt,
die zur effizienten bakteriellen Produktion und Reinigung von Proteinen
dienen. Hierzu dienen z.B. der besonders stringent regulierte
tet-Promotor für die stabile Produktion auch toxischer Proteine in
E. coli. Das Plasmid pTUM4 gestattet die Koexpression vier
periplasmatischer Faltungshelferproteine, was in vielen Fällen die
sekretorische Proteinproduktion in E. coli verbessert. Das Strep-tag
ist ein kurzes Peptidanhängsel mit neun Aminosäuren, welches
aufgrund seiner intrinsischen Affinität zu Streptavidin die
affinitätschromatographische Einschrittreinigung von rekombinanten
Proteinen (aus Extrakten von E. coli, Hefe oder Zellkulturen)
gestattet. Dieses System wurde mit kombinatorischen Verfahren und durch
Proteinstrukturanalyse über die Jahre verbessert und stellt heute
mit dem Strep-tag II und der StrepTactin-Affinitätsmatrix eine
robuste und weitverbreitete Methode für die Molekularbiologie und
Proteomforschung dar (Skerra & Schmidt, 2007). Zusammen mit
Reagenzien, die auch den Nachweis oder gar die stabile Immobilisierung
von Strep-tag(II)-Fusionsproteinen ermöglichen, wird dieses System
von der IBA GmbH vermarktet.
• Instrumentelle Methoden
- Molecular Modelling
- Molekularbiologie, Gensynthese, -sequenzierung
- Screening-Systeme: Phage Display, Colony Screening,
Fluorescence-activated
 Cell Sorting, Laborautomation Cell Sorting, Laborautomation
- Proteinproduktion in E. coli und in Zellkultur, Laborfermenter
- Proteinreinigung, Chromatographie
- Spektroskopie & Biophysik: UV/VIS, CD, Fluoreszenz (incl. Lebensdauer), SPR
 (Biacore), DLS, ITC, ESI-MS (Bruker maXis), Stopped Flow Kinetik (Biacore), DLS, ITC, ESI-MS (Bruker maXis), Stopped Flow Kinetik
- Röntgen-Strukturanalyse von Proteinen (Tecan-Kristallisierrobotik, Formulatrix
 RockImager, Rigaku-Röntgengenerator, Osmic-Optik, Mar345 Image Plate RockImager, Rigaku-Röntgengenerator, Osmic-Optik, Mar345 Image Plate
 Detector, Oxford Cryosystem) Detector, Oxford Cryosystem)
|